Rencontres et projets
Janvier 2018, je suis étudiante en 3ème année d’école d’ingénieur en agronomie (ENSAIA), spécialisée en biotechnologies. Tandis que je serai diplômée dès octobre, je souhaite poursuivre mes études par une thèse. Mais pas n’importe quelle thèse, je cherche un sujet suffisamment original complet pour m’inspirer et me donner l’envie d’y investir toutes mes compétences. Alors plutôt que de répondre à des appels d’offre qui ne m’inspiraient qu’à moitié, j’ai pris le problème dans l’autre sens, selon moi, le vrai sens, et je suis partie à la rencontre d’équipes de recherche qui travaillent sur des sujets qui me passionnent. Pour les contacter, j’envoyais un mail racontant qui j’étais, quel était mon parcours, ce que je cherche et leur demandant s’ils seraient disponibles pour une rencontre, afin d’échanger à propos de leurs recherches. Et bien sûr, mon but sous-jacent était de me créer un réseau de contact suffisamment vaste pour dénicher le poste qui me ferait vibrer. C’est dans ce contexte que j’ai rencontré Stéphane Lemaire, directeur de recherche et chef de l’équipe de recherche Biologie de Synthèse et des Systèmes des Microalgues au CNRS. Dans son équipe, pas de poste à pourvoir pour une doctorante, en revanche… une opportunité incroyable, et joliment amenée : apparemment mon CV est celui qu’on attendait pour lancer un projet hors du commun : stocker des données dans l’ADN, il faut bien reconnaitre qu’à ce moment-là, je suis flattée et carrément enthousiaste, vous vous en doutez : j’accepte.
Me voilà donc pas du tout en thèse mais ingénieur d’étude, seule personne travaillant à plein temps sur un projet hors du commun : encoder la Déclaration des Droits de l’Homme et du Citoyen (1789) et la Déclaration des Droits de la Femme et de la Citoyenne (1791) dans de l’ADN.
« Ok c’est bien gentil, on dirait qu’on parle d’un truc de fou, mais qu’est-ce que ça veut dire « encoder dans de l’ADN » en fait ? »
Qu’est-ce que l’ADN ?

L’ADN est une jolie molécule, en forme de double hélice, qui se trouve être le support de notre information génétique (c’est une sorte de bibliothèque dans laquelle est rangée chacun de nos gènes). ADN est un acronyme, pour Acide DésoxyRibonucléique, autrement dit : un acide auquel il manque un oxygène (« désoxy ») sur une molécule de sucre (le ribose) L’ADN varie d’un individu à un autre (surtout si ceux-ci n’appartiennent pas à la même espèce), en revanche il est le même pour chacune de nos cellules. Chaque cellule de notre corps lit le même ADN.
Mais du coup, vous allez me dire « Comment ça se fait que chacune de nos cellules lise la même information génétique, alors qu’on a plein de cellules différentes ? ». Effectivement, une cellule d’œil n’est pas une cellule de cuir chevelu, car on n’a jamais vu quelqu’un avoir des cheveux qui lui poussent dans les yeux. Et bien l’existence de types de cellules différentes au sein d’un même individu s’explique par le fait que chaque type de cellule lit un chapitre dédié de l’ADN, et non pas tous les chapitre. Une cellule d’œil lira donc le chapitre « Comment être une bonne cellule d’œil », mais ne lira pas le chapitre « Quels sont mes droits et mes devoirs en tant que cellule du cuir chevelu ».
« Ok, les cellules lisent un gros bouquin appelé ADN donc… Mais elles le lisent dans quelle langue ? ». Le langage génétique ne repose pas sur 26 lettres comme l’alphabet français ou anglais, mais juste de 4 lettres, chaque lettre désignant en fait une petite molécule appelée base de l’ADN : A pour adénine, T pour thymine, G pour guanine et C pour cytosine. (Petite précision: on a dit précédemment que l’ADN était une double hélice : aussi, en face de chaque base, se trouve une autre base qui lui fait face.
Cette correspondance base-base n’est pas hasardeuse : en face d’un A on trouvera toujours un T et en face d’un C toujours un G !). C’est la juxtaposition de ces lettres qui forme la molécule d’ADN. A certains endroits, cette juxtaposition de lettres formes un « mot génétique » qui a du sens pour la cellule, c’est-à-dire qu’en lisant cette partie là de l’ADN, la cellule sera capable de produire une protéine associée, comme si elle lisait le mode d’emploi de la construction d’un édifice de LEGO par exemple. Ces parties particulières de l’ADN sont appelées les gènes. Il y en a plusieurs répartis le long de l’ADN, séparés par de longues suites de A, T, G et C qui elles en revanche n’ont pas de signification particulière pour la cellule.

Que signifie « encoder dans de l’ADN » ?
L’ADN a donc un langage quaternaire, de 4 lettres. Les données numériques (par exemple, cet article de blog que vous êtes en train de lire) sont lues par l’ordinateur dans un langage binaire, constitué uniquement de 0 et de 1. Encoder des données numériques dans l’ADN, c’est donc trouver une correspondance entre le langage numérique et le langage génétique. Plusieurs correspondances ont été imaginées, par exemple :
– Exemple de correspondance compacte : A = 00, T = 11, G = 10, C = 01
Code employé par Georges Church, qui permet d’encoder un maximum d’information dans un minimum de bases d’ADN. Mais n’oublions pas que l’ADN que nous fabriquerons dans le cadre précis de notre projet va être mis dans un organisme vivant (une bactérie) pour sa duplication, il peut avoir un sens biologiquement parlant. Avec ce code-là, il n’existe qu’une seule traduction possible. Ce qui veut dire qui si par malchance (bien que ce soit assez imbrobable) la cellule peut comprendre cette séquence d’ADN et créer une protéine toxique (que ce soit pour l’expérimentateur ou la bactérie) on ne peut pas y remédier.
– Exemple de correspondance flexible : A = 0, C = 0, T = 1, G = 1
Code que j’ai employé au sein de l’équipe de Stéphane Lemaire, permettant d’encoder la même information numérique de plusieurs façons en langage génétique. J’expliquerai l’intérêt de cette méthode ci-après.
Toutes sortes de données peuvent être numériques. Par exemple, le texte de la Déclaration des Droits de l’Homme et du Citoyen peut être réécrit à l’ordinateur (format numérique de type texte) ou bien numérisé (format numérique de type image). Mais on peut aussi citer la musique ou encore les films par exemple.
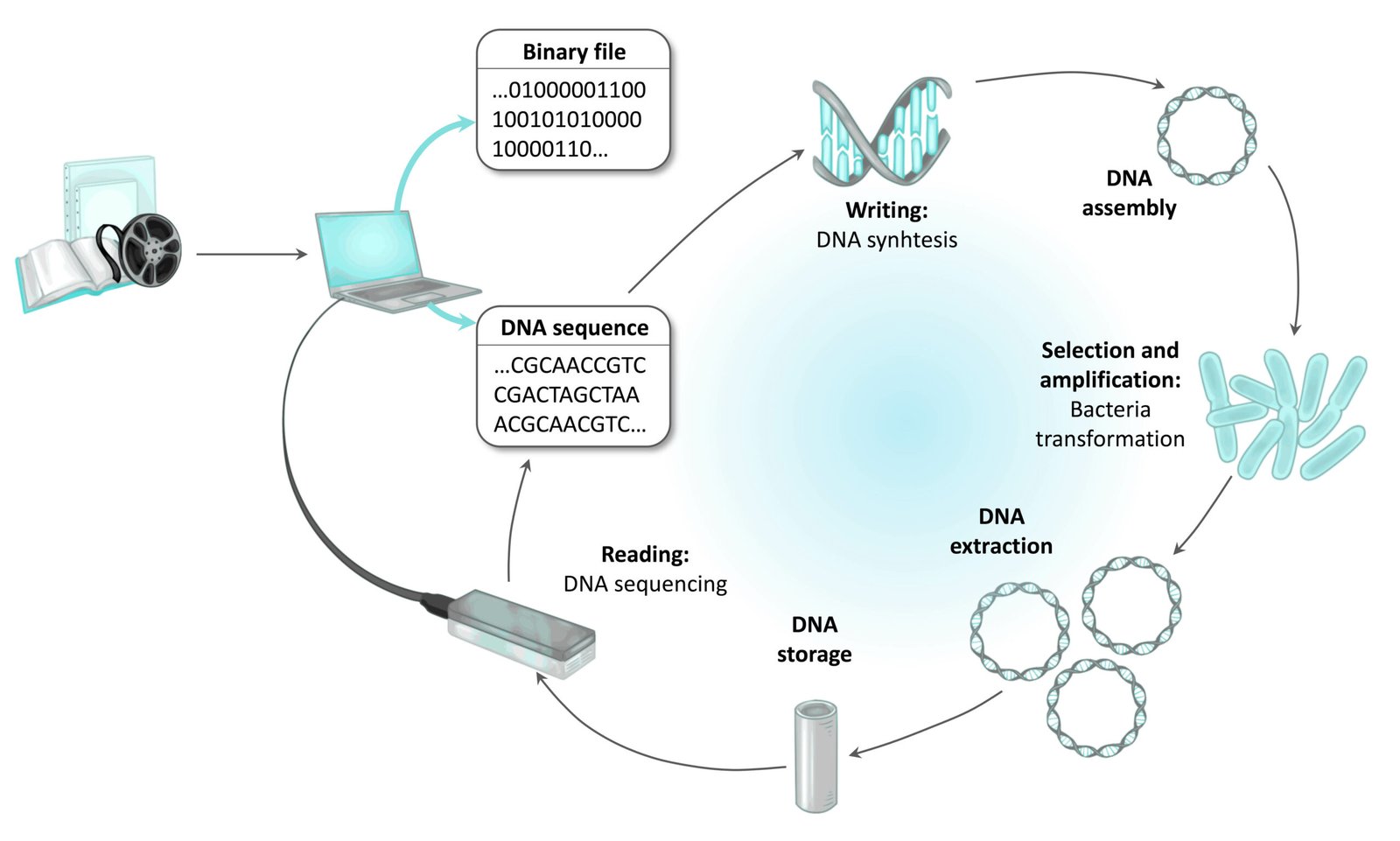
Une fois qu’on a la séquence génétique que nous avons générée en correspondance avec le fichier numérique que nous souhaitions coder, nous le faisons synthétiser. C’est-à-dire que nous envoyons la séquence de A, T, G, C à une entreprise de séquençage, qui s’occupe d’assembler les bases (molécules qui constitueront ce bout d’ADN) dans l’ordre indiqué. Twist Bioscience est une des entreprises dont l’activité est de synthétiser des bouts d’ADN par exemple. C’est d’ailleurs à eux que nous nous sommes adressés pour réaliser notre projet d’encodage de données dans l’ADN.

Les fragments d’ADN synthétisés sont ensuite assemblés chimiquement par des enzymes, dans des structures d’ADN circulaires appelées plasmides. Ce sont ces plasmides qui sont ensuite intégrés dans des bactéries, qui vont faire office de moines copistes redoutablement efficaces : elles vont multiplier ces ADN en des milliers de copies, et ce en l’espace de quelques heures seulement. Les plasmides ainsi amplifiés sont ensuite extraits des bactéries afin de s’affranchir de tout être vivant au moment du stockage. Puis il est lyophilisé et encapsulé, dans des capsules conçues spécialement pour stocker de l’ADN, technologie développée par Imagene.
Et si on souhaite lire son contenu ? On ouvre la capsule avec un décapsuleur, on dépose une goutte d’eau à l’intérieur pour remettre les molécules d’ADN en suspension, on en prélève une partie et on la dépose sur le lecteur d’un séquençeur d’ADN (nous avons utilisé le MinION développé par Nanopore et qui n’est pas plus grand qu’une clef USB, directemement relié à notre ordinateur !).
Grâce au programme informatique que nous avons développé, ces longues séquences de A, T, G et C sont ensuites retraduites en binaire avant d’être reconverties en texte (ou image selon le fichier de départ) lisible par l’oeil humain.
« Et voilà ! »
Pourquoi stocker de l’information numérique dans de l’ADN ?
On pourrait se dire que c’est juste de l’esbrouffe pour faire des trucs cools. Alors oui, c’est vrai que c’est très cool, mais en plus ça a un réel intérêt !
Aujourd’hui, comment stockons nous nos données numériques ?
Aujourd’hui, les données numériques que nous stockons (nos photos enregistrées sur notre Drive, nos musiques dans le Cloud, nos vidéos sur Internet, dont les réseaux sociaux, etc) sont hébergées dans des Data Centers, ou centres de données, qui sont de grands hangars renfermant des disques durs en permanence alimentés en électricité. Internet consomme donc en fait énormément d’énergie, rien que pour maintenir tous ces disques de stockage en perpétuel fonctionnement et dans les meilleures conditions de préservation possibles (température et humidité constantes, refroidissement des machines). En fait, si Internet était un pays, il ne serait rien de moins que le 3ème consommateur d’électricité au monde, tout juste derrière la Chine et les Etats-Unis.

Ces Data Centers présentent de plus l’inconvénient d’une importante emprise au sol. L’ensemble des Data Centers du monde occupe plus de 42 millions de m², soit 1.4 fois la taille de la Belgique entière, et cela sans produire, ni bière, ni frite, ni chocolat… Donc tout un espace qui ne peut être utilisé pour l’agriculture, les forêts ou le logement par exemple.
Et puis les disques durs utilisés actuellement pour stocker nos données numériques doivent être régulièrement changés, déjà parce qu’ils ont des dates de péremption assez courtes, mais aussi parce que les technologies changent (bandes magnétiques, disques SSD, etc…), générant autant de déchets.
En résumé, les méthodes de stockage des données numériques actuelles présentent les limites suivantes :
– elles consomment énormément d’énergie
– prennent de l’espace au sol considérable
– doivent être fréquemment renouvelées
Pourquoi l’ADN est un support de stockage super sexy
L’ADN est le support de l’information génétique depuis le début de la vie sur Terre. Cela fait donc presque 4 milliards d’années que ce support de stockage des données est mis à l’épreuve et perdure. Alors forcément, autant s’en inspirer pour nos propres stockages de données non ?
L’ADN humain contient environ 3.2 milliards de bases (pour rappel, une base est soit un A, un T, un G ou un C). En utilisant le système de correspondance compacte énoncé plus haut, ces 3.2 milliards de bases pourraient stocker 0.8 Go (giga octets) de données. Or, notre ADN on ne le voit même pas à l’oeil nu tellement il s’agit d’une molécule compacte ! Si bien qu’avec une telle technologie, on pourrait stocker l’entièreté d’Internet dans un volume pas plus grand qu’un litre.
(Parenthèse nerd :
Pour la séquence un peu nerd de cet article, voici un court déptail de la capacité de compaction du stockage ADN : avec une densité maximale de 4,5×1020 octets, soit 0,45 Zettabytes (ZB) équivalent à 450 x106 Terabytes (TB) par gramme d’ADN, toutes les données numériques de l’humanité (45 ZB) pourraient tenir dans 100 g d’ADN.
Fin de la parenthèse nerd)
Et les avantages ne s’arrêtent pas là ! Alors qu’on parvient à lire de l’ADN de mammouths à partir de vestiges figés dans la glace pendant des milliers d’années, a-t-on déjà réussi à lire un CD conservé dans de telles conditions, ne serait-ce que quelques années ? L’ADN est une molécule extrêmement stable dans le temps, conservable à température ambiante. Ce qu’il faut en revanche c’est la protéger des rayons UV, qui sont mutagènes (d’où l’utilité de la crème solaire que nous mettons sur notre peau !). Le stockage de l’ADN lui-même ne nécessite donc pas d’énergie puisque ces molécules sont parfaitement stables à température ambiante dans des conditions appropriées. La difficulté dans le fait de stocker des données dans le l’ADN ce n’est donc pas la stabilité de l’ADN en tant que telle mais celle du support sur lequel on va mettre cet ADN. Hé oui, parce qu’il faut bien le poser quelque part cet ADN ! Je vous révèlerai la méthode pour laquelle nous avons optée dans les prochaines lignes.
« Ok j’ai compris, c’est assez pratique de stocker des trucs dans l’ADN en fait. Mais du coup, pourquoi la Déclaration des Droits de l’Homme ? »
Encoder la Déclaration des Droits de l’Homme et du Citoyen… et celle de la Femme
Le projet de Stéphane Lemaire était que nous développions une méthode inédite de stockage dans l’ADN. Et pour prouver que cette méthode fonctionne, il fallait évidemment que nous choisissions quelque chose à encoder. L’idée était de montrer que l’ADN peut être le nouveau support d’archivage, et impossible de penser à l’archivage sans penser aux Archives Nationales. Nous souhaitions ce projet profondément français, en tant que première tentative de stockage dans l’ADN par une équipe française, et un des textes emblématiques de la France et de sa République, c’est bien la Déclaration de Droits de l’Homme et du Citoyen, de 1789. Et parce que notre projet est cependant moderne, ce texte ne pouvait exister sans son pendant féminin, la Déclaration des Droits de la Femme et de la Citoyenne, rédigée par Olympe de Gouges en 1791 (et puis étant la seule fille de l’équipe, il était quand même bien normal de rajouter un peu de droit des femmes dans tout ça non?).
Un projet d’une grande dimension philosophique et historique
Alors a commencé une grande période d’aller-retours dans les bibliothèques de Paris, à la recherche des textes originaux. Ceux que nous trouvons sur Internet sont en général corrigés pour convenir à notre langue française moderne. Or les textes originaux comprennent des lettres qui n’existent même plus aujourd’hui, comme le « s long », ainsi que des orthographes que nous jugerions fausses désormais. Alors la grande question était : faut-il conserver le texte réellement original, avec ses erreurs et ses s longs ? Mais qui suis-je pour y répondre ? Clairement pas la bonne personne.
Je suis alors partie à la rencontre de philosophes et historiens spécialistes dans les droits de l’homme et dans ceux de la femme, en réalisant un véritable travail de détective à la recherche des contacts des différentes personnes que je souhaitais faire prendre part au projet. J’ai en effet joui d’une très grande liberté d’action pendant mon année dans l’équipe de Stéphane Lemaire, me permettant d’entrer en contact direct avec qui je pensais opportun pour le développement de ce projet. J’ai ainsi eu la chance d’organiser des interviews privées avec Olivier Blanc, historien et conférencier spécialisé dans la Révolution française ; Geneviève Fraisse, philosophe française de la pensée féministe, directrice de recherche émérite au CNRS / CRAL – EHESS ; Marcel Gauchet, philosophe et historien, rédacteur en chef d’une des principales revue intellectuelles françaises, Le Débat chez Gallimard ; Maria Liouliou, spécialiste du patrimoine mondial au Centre du patrimoine mondial de l’UNESCO. Des rencontres à la fois essentielles à l’avancée de ce projet passionnant mais aussi extrêmement enrichissantes d’un point de vue personnel. (J’en profite d’ailleurs pour vous renouveller ma reconnaissance pour le temps et l’intérêt que vous m’avez accordés !)
Au final, les s longs n’ont pas été conservés pour cet encodage. En revanche, la Déclaration des Droits de la Femme et de la Citoyenne s’accompagne de plusieurs textes étonnants dans leur modernité et leur sarcasme (je vous recommande vivement de les lire ! Et je rédigerai très probablement des billets de blog à leur sujet pour vous donner la joie d’accéder à ces bijoux de l’histoire). Le texte original d’Olympe de Gouges contient : un préambule adressé aux femmes de tout âge et situations sociales pour les exhorter à se « réveiller » et à combattre les injustices liées à leur sexe ; une lettre à la reine, Marie-Antoinette (hé oui, directement ! Je fais pâle figure à côté à ne même pas avoir contacté le Président pour ce projet), l’enjoignant à oeuvrer pour son propre sexe ; et un postambule adressé à la gente masculine, commençant par l’intercation très forte « Homme, es-tu capable d’être juste ? ». Et juste pour le plaisir de son premier paragraphe, en voici un extrait, dont on ne peut qu’apprécier la modernité (et la déplorer en même temps, car le discours que nous tenons d’aujourd’hui fait en fait douloureusement échos à ce texte de 1791) :

Un projet alliant la biologie, la physique, la chimie et l’informatique

La biologie est une discipline qui a cette particularité de constamment nécessiter l’avancé d’autres disciplines scientifiques, en particulier la physique et la chimie. Des exemples pour mieux s’en rendre compte ? L’existence de microorganismes n’a pu être prouvée qu’en les observant au microscope, un outil constitué d’un ensemble de lentilles de verre, mis au point grâce à l’optique, une discipline issue de la physique.
Plus précisément, dans notre cas particulier d’encodage de données numériques dans l’ADN, l’étape de biologie est encadrée par une étape de chimie en amont et de physique en aval, sans oublier les étapes relevant du programme informatique créé.
– De l’informatique : pour traduire le langage binaire en quaternaire.
– De la chimie : pour synthétiser les séquences d’ADN que nous souhaitons stocker. Cette étape est malheureusement – pour le moment – dépendante de la pétrochimie. Mais des recherches existent pour développer de nouvelles méthodes de synthèse d’ADN qui soient plus vertes (rejoignez donc l’aventure de la recherche dans le domaine !). Comme les technologies de séquençage actuelles ne permettent pas de synthétiser de très longs fragments d’ADN, une autre étape de chimie est essentielle : l’assemblage ex vivo par des enzymes, des fragments d’ADN synthétiques dans des structures ADN (des palsmides) capables d’être reconnues par la bactérie.
– De la biologie : car nous utilisons des bactéries pour multiplier en très grand nombres de copies les séquences d’ADN assemblées. L’ADN étant ensuite extrait des bactéries, il n’y a plus aucune étape liée à la biologie dans la suite du processus, puisque l’ADN est une molécule comme une autre une fois sortie de son contexte vivant.
– De la physique : pour séquencer l’ADN, c’est à dire le lire, donc déterminer l’ordre des bases d’ADN A, T, G et C.
– De l’informatique : encore ! Pour traduire la séquence dans l’autre sens dans l’autre sens.
L’encodage de données numériques dans l’ADN est pour le moment réservé au stockage froid, c’est-à-dire de l’archivage de données qui sont très rarement relues ou modifées. Et on ne s’en rend pas compte, mais le stockage froid représente 85% du stockage total ! Prenez par exemple les actes notariaux : personne de les relit, et pourtant il est obligatoire de les conserver pendant plusieurs année. Mais si des percées sont faites dans le domaine de la physique par exemple, on pourrait imaginer séquencer l’ADN instantannément et sans erreur, permettant d’élargir l’application du stockage numérique dans l’ADN à du stockage de données relues fréquemment !
Et la suite de cette grande aventure, c’est quoi ?
Remise des capsules d’ADN aux Archives Nationales : une première mondiale

Une des grandes attentes pour nous concernant ce projet était le dépôt des capsules contenant les molécules d’ADN dans lesquelles sont stockées les Déclarations des Droits de l’Homme et du Citoyen et de la Femme et de la Citoyenne aux côtés du texte original de la Déclaration des Droits de l’Homme et du Citoyen, conservé dans la Grande Armoire de Fer des Archives Nationales de Paris. La recherche étant ce qu’elle est, particulièrement lorsqu’on travaille avec du vivant, nous avons du reculer la date de notre visite aux Archives Nationales d’un an.
C’est donc en novembre 2021 que nous avons été conviés à cette conférence de presse d’envergure nationale, accompagnés de multiples journalistes représentants de stations de radio telles que France Culture, Le Monde, France Inter, etc.

Création de Biomemory pour valoriser le brevet d’encodage de données numériques dans l’ADN
La technologie que nous avons développée à cinq, avec Stéphane Lemaire, Pierre Crozet, Alexandre Maes et Zhou Xu, ayant donné lieu à un brevet, Stéphane Lemaire et Pierre Crozet ont co-créé Biomemory pour valoriser cette technologie. Biomemory est une start-up française spécialisée dans l’encodage de données numériques dans l’ADN et qui valorise la technologie brevetée. Ils ont été rejoint par Erfane Arwani, CEO et cofondateur de Biomemory.

… et je profiterai d’un prochain numéro de blog pour vous détailler la suite des aventures de Biomemory, ainsi que pour vous donner de plus amples détails sur cette technologie passionnante qu’est le stockage de données numériques dans l’ADN !
Et pour finir, dans le cadre de la communication de Biomemory auprès de ses clients et investisseurs, j’ai créé pour Biomemory, par l’intermédiaire de Beink, un trophée contenant deux capsules d’ADN : l’une pour la Déclaration des Droits de l’Homme et du Citoyen, l’autre pour la Déclaration des Droits de la Femme et de la Citoyenne, avec en fond un dessin réalisé pour l’occasion, retraçant l’histoire de ces textes, depuis Révolution jusqu’à leur encodage dans l’ADN en laboratoire. Un sacré périple !

N’hésitez pas à soutenir Beink en partageant ses articles ou en vous inscrivant à sa newsletter